科研进展 | 基于结构的蛋白质-小分子评分函数设计
近期,数字所魏彦杰研究员课题组在国际知名生物信息学期刊Briefings in Bioinformatics(影响因子IF=11.622)上发表了一篇题为“Improving protein–ligand docking and screening accuracies byincorporating a scoring function correction term”的论文,提供了一种全新的蛋白质-小分子评分函数的设计思路,即结合对小分子对接构象的偏差估计作为一种传统打分函数的修正项,可以显著提升分子对接和筛选的精度,对于小分子药物设计和筛选有着重要意义。论文通讯作者为数字所魏彦杰研究员和慕宇光教授(新加坡南洋理工大学),论文第一作者为郑良振博士。
评分函数(scoring function)是蛋白质-小分子对接和筛选中的关键要素。随着蛋白质-小分子结构和亲和力数据库的持续扩增,让机器学习和深度学习也逐渐在评分函数设计上有了比较好的应用空间。虽然已有多种基于高度精确的深度学习或机器学习的评分功能但它们在对接和筛选方面的直接应用比较有限。作者提出了OnionNet-SFCT模型,关注小分子的对接构象和真实构象之间的偏差,并可结合传统打分函数用于多种小分子相关应用场景。模型通过显性的原子-氨基酸相互作用描述,使用基于Adaboost的随机森林模型,来拟合蛋白质-小分子的结合模式和真实状态之间的偏差(RMSD)。
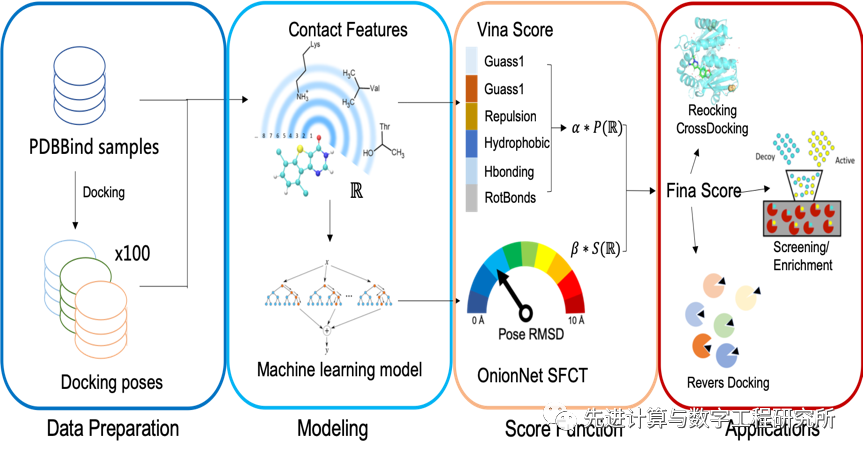
图1. OnionNet-SFCT+Vina评分模型的设计和应用
作者发现现有模型对于近真实状态(near-native)的偏差预测不如传统算法,但对于整体大范围的构象偏差预测则更准确,因此该模型可以很好地和传统方法进行整合,来作为一种有益的修正。单独使用OnionNet-SFCT评分能量,不能显著提高重对接性能,然而如果将OnionNet-SFCT作为传统对接评分函数的能量修正项,可以在多个数据集上提升分子对接的成功率。例如,OnionNet-SFCT修正Vina之后,可以在交叉对接任务上将top1构象的RMSD平均降低0.736倍,且可将top1构象的成功率提升10.6%(图2)。
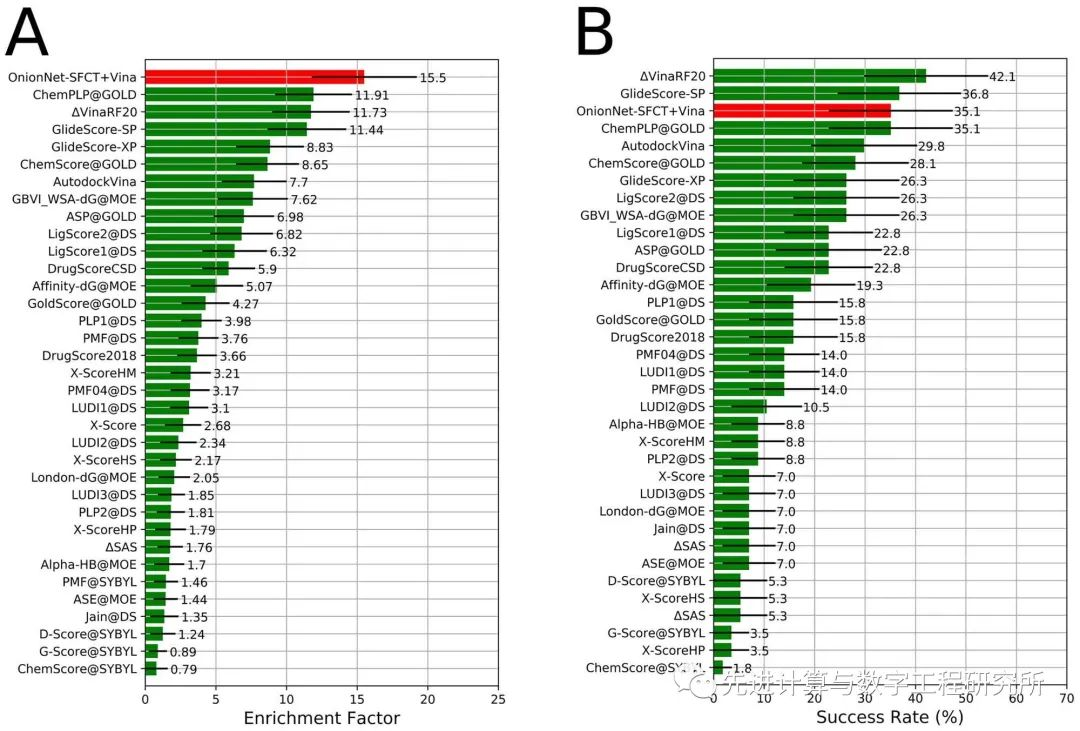
图2.模型在CASF-2016数据集上的表现
另外,结果表明OnionNet-SFCT修正后的Vina评分函数或OnionNet-SFCT修正Gnina的评分函数对于筛选任务更为准确。相比于重对接和交叉对接任务的性能提升,在DUD-E和DUD-AD数据集的筛选能力的关键指标富集因子(EF1%)性能提升更为显著(图3)。
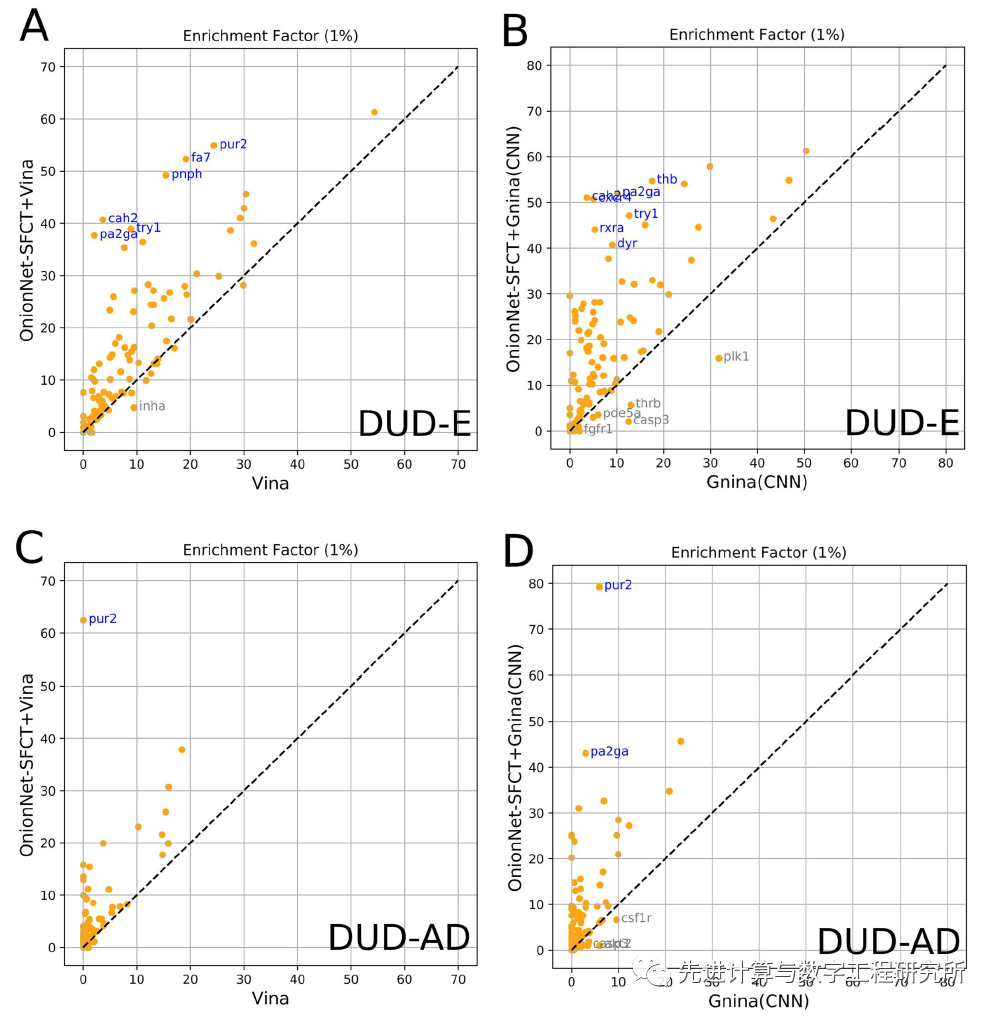
图3. OnionNet-SFCT修正在DUD-E和DUD-AD上筛选效果和修正前评分函数的比较
作者提出的模型可以与多个对接应用程序相结合,以提高小分子构象选择的准确性和筛选能力,可以广泛用于基于结构的药物发现,有助于基于结构的药物发现和未来潜在的靶点寻找。
